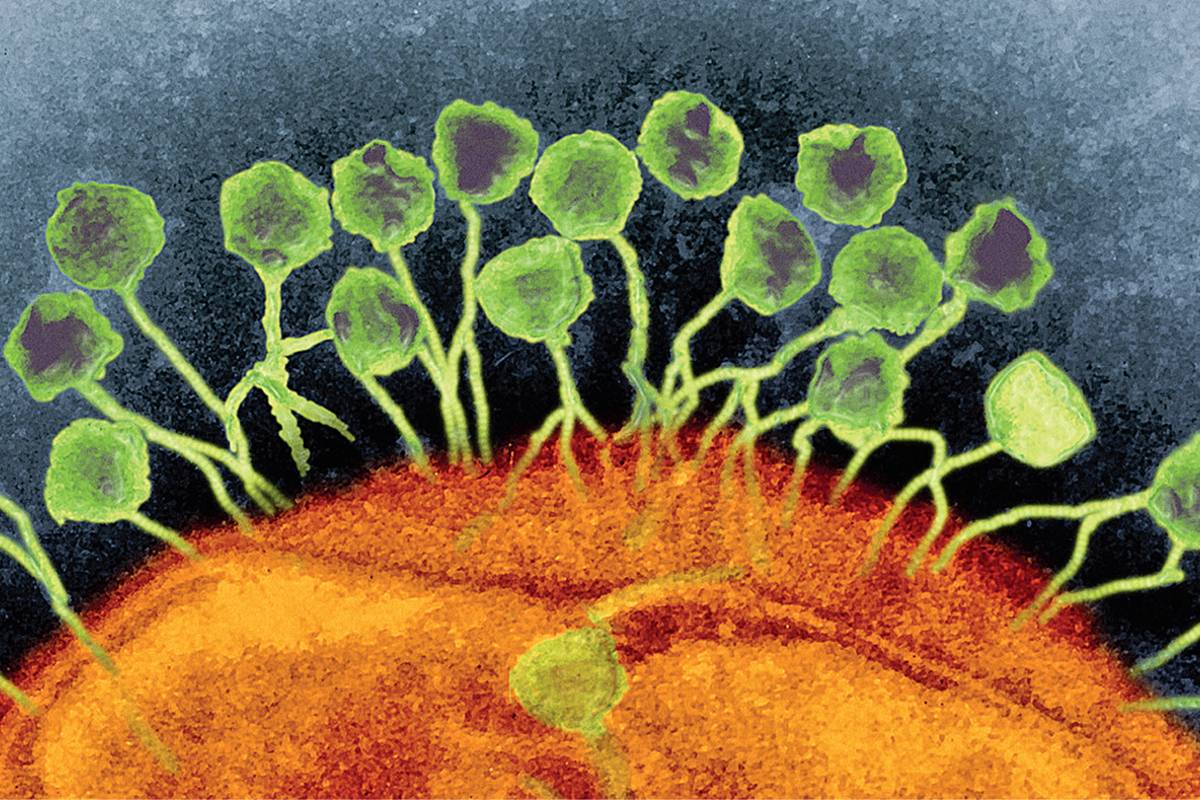
Desde hace unos años, el microbioma intestinal humano -el conjunto de microorganismos que viven en nuestro intestino- se ha convertido en diana de muchos investigadores por su repercusión tanto en la salud como en la enfermedad. No solo ayuda a digerir los alimentos, sino que protege contra las bacterias patógenas, modula el bienestar mental y prepara y regula el sistema inmunológico, entre otras muchas funciones, pero en ocasiones su desregulación por estrés oxidativo o toxinas bacterianas puede conducir a la llamada disbiosis. Aunque es uno de los ecosistemas microbianos más estudiados, más del 70 por ciento de las especies microbianas que viven allí aún no se han cultivado en el laboratorio.
En el último número de Nature Microbiology, un equipo de las universidades de Stanford, en California, y de Queensland, en Australia, y del Joint Genome Institute de California, recoge la identificación de 54.118 especies de virus que viven en el intestino humano, el 92 por ciento de las cuales eran desconocidas previamente. Para ello se han servido de la metagenómica, una técnica mediante la cual el ADN se extrae directamente de un entorno y se secuencia aleatoriamente, proporcionando una instantánea de lo que está presente en su interior y de lo que podría estar haciendo.
Según explican en The Conversation Philip Hugenholtz y Jen Low, de la Universidad de Queensland, dos de los firmantes del estudio, para su catálogo -el Metagenomic Gut Virus- extrajeron secuencias virales de 11.810 metagenomas fecales disponibles públicamente, tomados de personas en 24 países diferentes. Así encontraron 189.680 genomas virales que representan más de 50.000 especies distintas de virus que en conjunto codifican más de 450.000 proteínas, “un enorme reservorio que puede ser beneficioso o perjudicial para sus huéspedes microbianos y humanos”.
Los reyes del intestino
El principal hallazgo es la confirmación de que la gran mayoría de esos virus -más del 75%- son bacteriófagos o fagos, ‘comedores de bacterias’, en especial contra las clases Bacteroidia y Clostridia, e inofensivos para las células humanas. Pueden afectar a los procesos del ecosistema microbiano a través de la depredación, la lisogenia y la transferencia horizontal de genes.
“Cuando pensamos en virus -explican los autores-, pensamos en organismos que infectan nuestras células con enfermedades como las paperas, el sarampión o, más recientemente, la covid-19. Sin embargo, hay un gran número de estos parásitos microscópicos en nuestros cuerpos -sobre todo en nuestro intestino- que se dirigen a los microbios que viven allí”.
En su rastreo metagenómico hallaron algunos patrones geográficos sorprendentes. “Por ejemplo, una subespecie del enigmático crAssphage, recientemente descrito, era frecuente en Asia, pero muy rara o ausente en muestras de Europa y de Norteamérica, un modelo de expansión localizada”.
Analizaron también las funciones de los retroelementos generadores de diversidad (DGR), una clase de elementos genéticos que mutan genes diana específicos con el fin de generar variaciones que puedan ser beneficiosas para el huésped. “En los virus, estos DGR pueden ayudar en la carrera armamentista evolutiva con sus huéspedes bacterianos”.
Observaron además que un tercio de las proteínas codificadas más comunes tienen funciones desconocidas, incluidos más de 11.000 genes relacionados con las beta-lactamasas, que contribuyen a la resistencia a antibióticos como la penicilina.
Defensores de agresiones
En otra fase del trabajo vincularon los fagos a sus huéspedes microbianos. Vieron que representan casi el 40% de los espaciadores CRISPR que se encuentran en las bacterias y arqueas del intestino humano. Los CRISPR, más conocidos por sus muchas aplicaciones en la edición de genes, son sistemas inmunes bacterianos que ‘recuerdan’ infecciones virales pasadas y evitan que vuelvan a ocurrir, una especie de autovacunas. Lo hacen copiando y almacenando fragmentos del virus invasor en sus propios genomas, que luego se pueden usar para atacar y destruir específicamente el virus en futuros encuentros. Es decir, estas secuencias repetidas en el ADN de las bacterias contienen el material genético de los virus que han atacado a las bacterias en el pasado, y que por eso permiten reconocer si se repite la infección y defenderse ante ella cortando el ADN de los invasores, como tijeras moleculares.
Como era de esperar, las especies virales altamente abundantes se vincularon a especies bacterianas también muy abundantes en el intestino, principalmente pertenecientes a los filos Firmicutes y Bacteroidetes.
Una aplicación prometedora de un inventario de virus intestinales y sus huéspedes sería la terapia con fagos, un concepto anterior incluso a los antibióticos, en el que los virus se utilizan para atacar selectivamente patógenos bacterianos con el fin de tratar infecciones.
Asimismo, puede ayudar a la personalización del microbioma mediante intervenciones dietéticas, probióticos, prebióticos o incluso los conocidos trasplantes de microbiota fecal, usados sobre todo contra el patógeno bacteriano Clostridium difficile, una de las principales causas de diarrea adquirida en el hospital que podría controlarse mediante fagos.
Varios catálogos
El catálogo Metagenomic Gut Virus (MGV) se une ahora y complementa a otros ya existentes como el Human Virome Database (HuVirDB), de la Universidad de California en San Francisco, el Gut Virome Database (GVD), de la Universidad Estatal de Ohio, con unos 142.000 genomas virales, y el Integrated Microbial Genome/Virus (IMG/VR), también del Joint Genome Institute.
Las diferencias entre estos catálogos virales se deben a factores como los conjuntos de datos utilizados para la extracción de metagenomas, los métodos para la identificación viral y los criterios para la inclusión de secuencias. Por ejemplo, el MGV mejora enormemente la cobertura de Microviridae que fueron excluidos del GVD debido a su corta longitud. En el futuro, estos y otros catálogos de genomas virales a gran escala podrían integrarse para crear un recurso unificado y estandarizado, como se realizó recientemente para los catálogos de genomas bacterianos del intestino humano.
Un análisis metagenómico dirigido desde el Joint Genomic Institute de California ha generado una monumental base de datos con los virus intestinales. Off José R. Zárate Microbiología y Enfermedades Infecciosas Offvia Noticias de diariomedico.... https://ift.tt/3w4chGL


No hay comentarios:
Publicar un comentario