Un nuevo método diseñado por científicos de la Universidad de Drexel, en Filadelfia, identifica y etiqueta las variantes mutadas del coronavirus SARS-CoV-2 con agilidad y eficacia.
Un primer análisis realizado con esta nueva herramienta, que se describe en PLOS Computational Biology, de una base de datos mundial sugiere que hay al menos entre 8 y 14 variantes ligeramente diferentes del virus circulando en Estados Unidos.
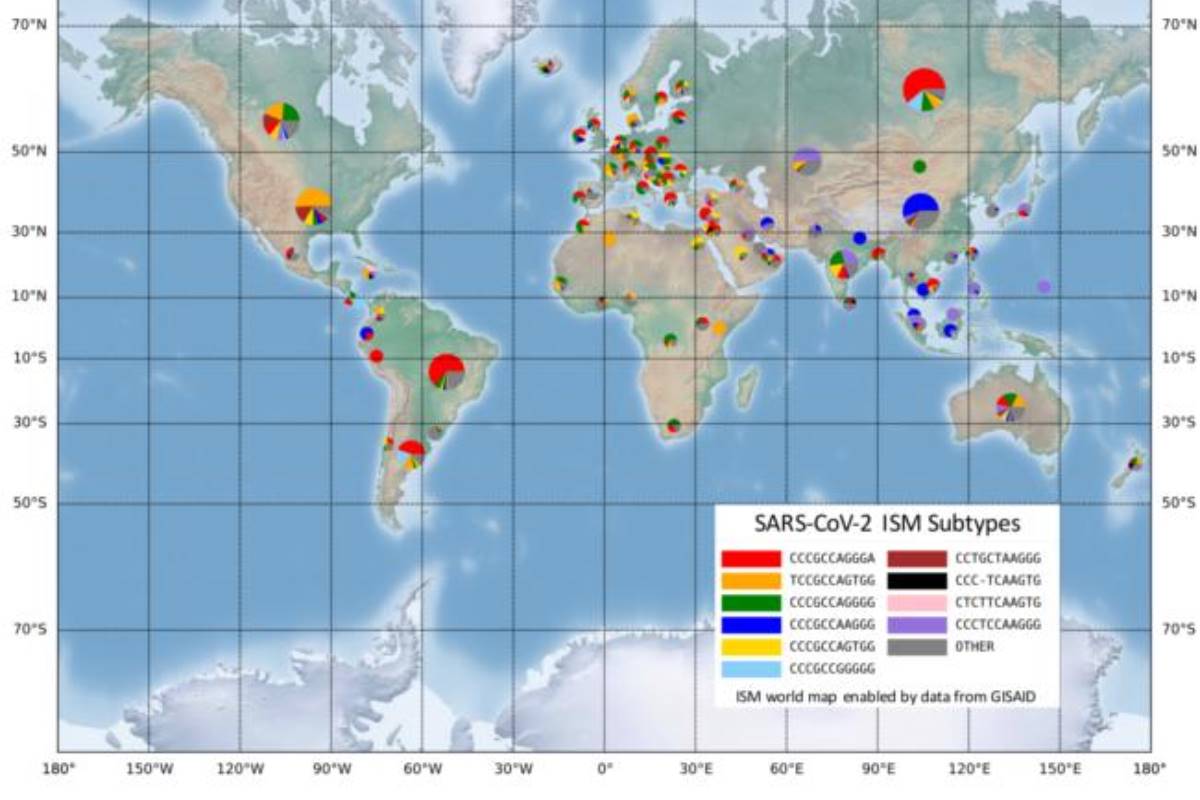
Algunas de esas variantes son iguales o han evolucionado de cepas asiáticas, y otras son las mismas que se encuentran en Europa. "Identificar las variaciones nos permite ver cómo ha cambiado el virus a medida que viaja de una población a otra. También puede mostrarnos las áreas donde el distanciamiento social ha logrado aislar a la covid-19”, afirma Gail Rosen, profesora en la Escuela de Ingeniería de Drexel y responsable del desarrollo de la herramienta.
Según Rosen, este análisis preliminar, utilizando información disponible en bases de datos públicas mundiales, “muestra que la combinación de subtipos de virus encontrados en Nueva York es muy similar a los encontrados en Austria, Francia y Europa Central, pero no en Italia. Y el subtipo de Asia, que se detectó aquí [Estados Unidos] en los momentos iniciales de la pandemia no se ha extendido mucho. En cambio, vemos un nuevo subtipo que solo existe en Estados Unidos como el más frecuente en el estado de Washington y en la costa oeste".
De acuerdo a esta investigación, desde principios de abril hasta finales del verano, mutaron a la vez tres posiciones genómicas en la secuencia del SARS-CoV-2. Una de ellas está asociada a la señalización y replicación celular. Otra, parece estar asociada con la formación de la proteína “Spike’, la parte del virus que permite su entrada en las células sanas; la tercera posición parece que no se traduce en proteína.
"Estamos viendo que la proteína de pico y la parte del virus responsable de empaquetar su material genético han desarrollado algunas mutaciones importantes, pero por lo demás están cambiando a un ritmo más lento", asegura Bahrad Sokhansanj, investigador en Drexel y otro de los autores del estudio.
Esos cambios, que aún deben ser investigados en mayor profundidad para determinar su impacto en la transmisibilidad y la patogenicidad del SARS-CoV-2, pueden servir para trazar una huella digital del patógeno que facilite la identificación de las diferentes cepas.
Gracias a la nueva herramienta no se requiere el análisis de la secuencia genética completa del virus para identificar sus mutaciones. En el caso del SARS-CoV-2, esto significa reducir el código genético del virus de 30.000 bases a una etiqueta de 20 bases.
"Es el equivalente a escanear un código de barras en lugar de escribir el número completo del código del producto", explica Rosen.
“Y ahora mismo, todos estamos tratando de pasar por la tienda un poco más rápido”, continúa Rosen con la metáfora. “Para los científicos, eso significa poder pasar a un análisis de nivel superior mucho más rápido”. Por ejemplo, puede acelerar el estudio sobre las diferentes versiones del virus y su relación con resultados clínicos. O permitir rastrear si los nuevos casos son el resultado de una transmisión local o provienen de otras regiones, ya sea nacionales o de otras partes del mundo.
De esta forma, se pueden alcanzar mejor objetivos clave, como “comprender la respuesta inmune del organismo, así como para identificar terapias antivirales y diseñar vacunas", apunta Sokhansanj.
via Noticias de diariomedico.... https://ift.tt/3ldMOFS


No hay comentarios:
Publicar un comentario