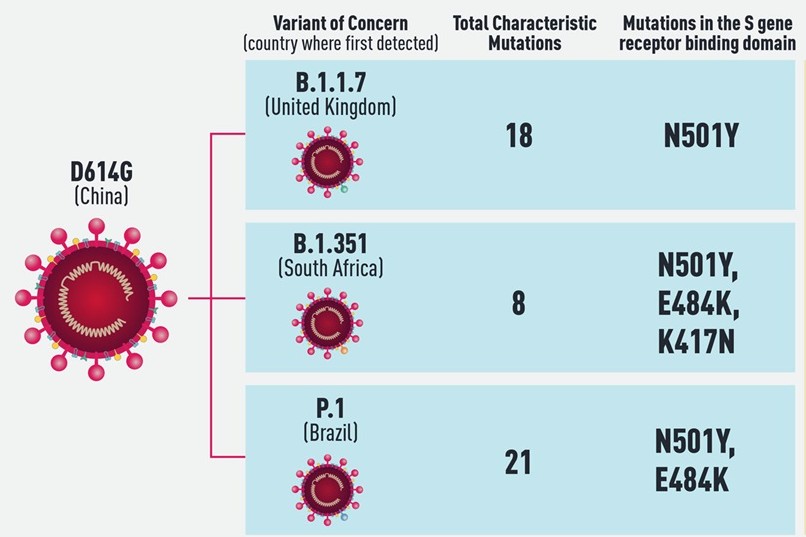
A la variante británica se la conoce como 20I/501Y.V1, VOC 202012/01 o B.1.1.7; a la sudafricana, como 20H/501Y.V2 o B.1.351; y a la brasileña, con 17 mutaciones exclusivas, como P.1, 20J/501Y.V3 y B.1.1.28. La mutación específica D614G, que desde hace unos meses se ha impuesto a la 614D, la comparten estas tres variantes.
¿A qué se debe tal batiburrillo de números e iniciales? «Para el VIH ya se han consensuado tipos, grupos, subtipos, subsubtipos y recombinantes ―explica África Holguín, responsable del Laboratorio de Epidemiología Molecular del VIH-1 del Instituto Ramón y Cajal de Investigación Sanitaria (Irycis), en el Hospital Ramón y Cajal de Madrid―, pero con las variantes del SARS-CoV-2 no hay unanimidad y sí varias entidades que las clasifican cada una a su manera».
Hasta diciembre de 2020, la iniciativa de ciencia global GISAID había identificado siete clados: O, S, L, V, G, GH y GR. También hasta diciembre de 2020, la plataforma suizo-estadounidense NextStrain había clasificado cinco: 19A, 19B, 20A, 20B y 20C. En noviembre pasado, un equipo de la Universidad Rey Abdalá, de Arabia Saudí, publicó en International Journal of Infectious Diseases cinco clados globales: G614, S84, V251, I378 y D392. El equipo de Andrew Rambaut, de Biología Evolucionista de la Universidad de Edimburgo, propuso el año pasado en Nature Microbiology que se empleen varios linajes para los subtipos de SARS-CoV-2, que en febrero de este año se han actualizado a seis: A, B, B.1, B.1.1, B.1.177, B.1.1.7 (el B.1 ha sido subdividido en más de 70 sublinajes).
Hay también un cluster 5 definido por el Instituto Estatal de Serología (SSI) de Dinamarca tras las mutaciones aparecidas en granjas danesas de visones. No hace mucho han aparecido las variantes B.1.429/CAL.20C, de California, la B.1.525 o VUI-202102/03, de Nigeria (que ya le habían adjudicado la B.1.1.207), y hay otra en estudio en Nueva York, perteneciente al linaje B.1.526 (o clado 20C de la clasificación de NextStrain). Y el activo equipo de Federico Martinón, de la Universidad de Santiago de Compostela, ha seleccionado cinco linajes principales en nuestro país: A2a5, B3a, B9, A2a4 y A2a10.
Clados, linajes, mutaciones, variantes, cepas… Bastante incomprensible, no ya para los profanos, sino incluso para los expertos. ¿Hay límites definidos? ¿Cuántas mutaciones se requieren para pasar de una a otra escala de diferenciación? ¿Qué porcentajes de homología genética marcan las fronteras? En el magnífico Glosario de covid-19 inglés-español de Tremédica y Cosnautas, que cuenta con la participación de Fernando Navarro, coordinador del «Laboratorio del lenguaje» en Diario Médico, especifican que, «en virología, los términos isolate, mutant, strain, type y variant se usan a menudo de manera laxa como si fueran sinónimos o intercambiables».
La Organización Mundial de la Salud (OMS), en una de sus últimas reuniones, ha dicho que está trabajando en una clasificación homogénea, pero que no quiere denominaciones referidas a países para no estigmatizar a nadie. La intención es buena, pero con los antecedentes que existen no será fácil una clasificación digerible. Al fin y al cabo, y pese a las alusiones nacionales, lo más claro ―y lo más empleado― son «las variantes británica, sudafricana, brasileña y demás». Aunque nos pueda molestar y sabiendo que no se originó en nuestro país, en todo el mundo se conoce a la gripe de 1918 como «gripe española».
José Ramón Zárate
reproducido a partir de «El SARS-CoV-2 muta diez veces menos que cualquier otro virus de ARN», en Diario Médico
via Noticias de diariomedico.... https://ift.tt/3kNRi74


No hay comentarios:
Publicar un comentario