El 14 de abril de 2003 es una fecha importante para la historia de la ciencia. Aquel día de hace poco más de 20 años se anunció el fin del Proyecto Genoma Humano: la secuencia esencial de nuestro ADN había conseguido descifrarse después de muchos años de esfuerzo.
No obstante, ese 'mapa', que costó 3.000 millones de dólares y que comenzó a usarse como referencia del genoma humano, no estaba completo. Tenía lagunas en varias regiones genéticas y se basaba principalmente en el ADN de unos pocos individuos de origen europeo. Y aunque en estas dos décadas se han producido avances tecnológicos que han permitido 'cartografiar' esas lagunas -la secuencia completa se obtuvo en 2022- y abaratar el proceso, seguía faltando una referencia que fuera más global y diversa.
A partir de ahora, gracias a un consorcio internacional financiado por el Instituto de Investigación Nacional del Genoma Humano de EEUU, ese recurso -un pangenoma de referencia- estará disponible.
El nuevo 'mapa', que se presenta como un primer borrador, incluye la secuencia genética completa de 47 individuos de diferentes orígenes, lo que proporciona información detallada de 94 genomas debido a que cada individuo lleva en su ADN dos copias de genes 'heredados' de su padre y su madre. El objetivo del proyecto es seguir añadiendo datos al 'mapa', por lo que prevé que a mediados de 2024 incluya información genética de 350 personas de ascendencia étnica diversa.
"Hasta ahora, la referencia que usaba la comunidad científica estaba incompleta y carecía de diversidad", señaló en rueda de prensa Benedict Paten, director asociado del Instituto de Genómica Santa Cruz de la Universidad de California y uno de los líderes del proyecto. Este nuevo recurso, en cambio, proporciona una imagen más completa y permitirá realizar análisis más certeros a la hora de caracterizar la variabilidad genética de la población humana sea cual sea su origen, destacó.
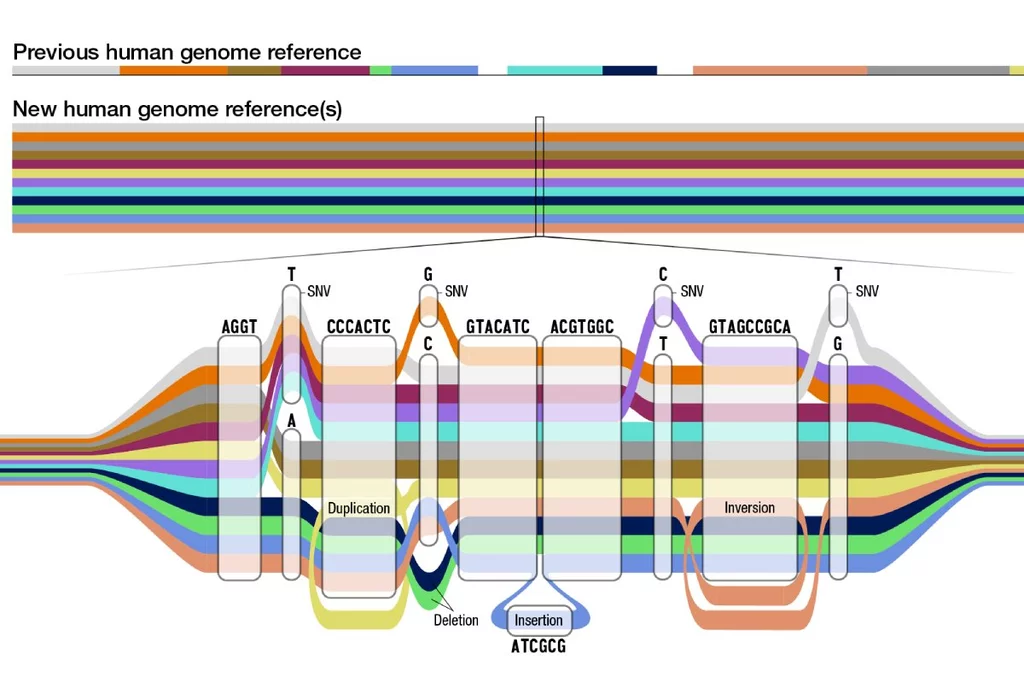
De hecho, el nuevo pangenoma ya ha sacado a la luz más de 100 millones de nuevas bases -cada una de las letras que componen el genoma-, y ha destapado nuevos alelos en regiones estructuralmente complejas del genoma que hasta ahora no figuraban en el genoma de referencia. Los detalles de la investigación se publican en cuatro artículos en el último número de las revistas Nature y Nature Biotechnology.
Mediante técnicas computacionales de última generación, los investigadores han sido capaces de construir un recurso que, en lugar de ser único y lineal, como era hasta ahora la referencia GRCh38 que se utilizaba, aporta distintas versiones de una misma secuencia al mismo tiempo, lo que proporciona a los investigadores un mayor abanico de opciones para sus análisis. En el proyecto ha participado un equipo del Centro de Supercomputación de Barcelona liderado por Santiago Marco-Sola.
Qué supone un nuevo pangenoma para la investigación
"Hasta ahora nos hemos contentado con una sola secuencia del genoma que en su día se decidió arbitrariamente que era la secuencia referencia, formada por trozos de secuencia de un puñado de personas de ascendencia principalmente europea. Y si bien esto ha tenido una gran utilidad, también tiene muchas limitaciones", señala Jorge Ferrer, investigador del Centro de Regulación Genómica de Barcelona (CRG). "Por ejemplo, aunque resulte sorprendente, a cada uno de nosotros nos pueden faltar o sobrar unos cuantos trozos muy grandes del genoma. Si el pedazo de genoma escogido para ser la referencia es de alguien que no tiene ese trozo (o lo tiene suficientemente alterado), el mapa de referencia que utilizamos actualmente no serviría para una persona que tiene una mutación que afecta esa parte", aclara. Para complicar aún más las cosas, continúa, "el genoma puede variar enormemente en diferentes partes del mundo. Y si el mapa de referencia está hecho con variantes europeas, tiene menor utilidad para interpretar el genoma de una persona de Camerún o China".
El trabajo actual, apunta Ferrer, "es el primer paso para resolver estos problemas". "Han creado un sistema complejo que permite cotejar la secuencia genómica de una persona con todas estas posibles secuencias humanas, en lugar de con una sola secuencia y el consorcio tiene planes de desarrollar esta estrategia con la secuencia de muchos más individuos".
Un recurso para la medicina
Para José Manuel Castro Tubío, líder del Grupo de Investigación de Genomas y Enfermedad del Centro de Investigación en Medicina Molecular y Enfermedades Crónicas (CIMUS) de Santiago, este nuevo recurso va a ayudar, en primer lugar, a "conocer mejor nuestra identidad, a conocer qué es lo que nos hace diferentes genéticamente a unos de otros". Y el hecho de "conocer lo que nos hace diferentes, qué secuencias de material genético nos hacen diferentes, nos va a permitir saber cosas acerca de nuestra evolución y nos va a permitir también saber cosas sobre las enfermedades genéticas que nos afectan".
"La variabilidad genética va asociada a rasgos biológicos y también a la predisposición de desarrollar enfermedades", explica. "Estos nuevos genomas que ahora se publican van a permitir descubrir muchas variantes que todavía no sabemos a qué se asocian".
"Toda la gente que está secuenciando ahora mismo genomas va a poder comparar las secuencias con estos genomas de referencia que están muy bien caracterizados. Y eso nos va a dar mucha más información de la que conseguíamos hasta ahora con el genoma humano de referencia que se consiguió en los primeros años 2000", subraya el investigador, quien remarca que el nuevo recurso supone "un salto cuantitativo más que cualitativo".
via Noticias de diariomedico.... https://ift.tt/dsAgkGV


No hay comentarios:
Publicar un comentario